Diese Seite wurde maschinell übersetzt.
Füllen Sie bitte eine 1-Minuten-Befragung zur Qualität dieser Übersetzung aus.
Entwicklung eines Deep-Learning-Modells für die assistierte Pathologiediagnose
Von Ong Kok Haur, Laurent Gole, Huo Xinmi, Li Longjie, Lu Haoda, Yuen Cheng Xiang, Aisha Peng und Yu Weimiao, Bioinformatics Institute
Prostatakrebs ist die zweithäufigste Form der Erkrankung bei Männern und wird üblicherweise durch die Untersuchung von Gewebeproben diagnostiziert. Dieser Prozess, der traditionell von erfahrenen Pathologen unter dem Mikroskop durchgeführt wird, ist arbeitsintensiv und zeitaufwändig. Darüber hinaus ist in vielen Ländern die Zahl der medizinischen Fachkräfte, etwa Pathologen, die solche Untersuchungen durchführen können, begrenzt, insbesondere bei hoher klinischer Arbeitsbelastung. Dies kann zu einem Rückstau von zu analysierenden Proben und zu Verzögerungen beim Behandlungsbeginn führen.
Teilweise aufgrund der Einschränkungen, die bei der manuellen Analyse von Proben auftreten, hat die Forschung zur Nutzung von KI und Deep Learning zur Unterstützung der pathologischen Diagnose von Prostatakrebs und anderen Krebsarten rasch zugenommen. Bevor Deep-Learning-Modelle entwickelt, optimiert, validiert und für klinische Anwendungen eingesetzt werden können, müssen jedoch mehrere technische Hürden überwunden werden. Schätzungsweise weisen beispielsweise etwa 15 % der digitalen Pathologiebilder Qualitätsprobleme auf, die unter anderem mit Fokus, Sättigung und Artefakten zusammenhängen. Darüber hinaus lässt sich die Bildqualität mit bloßem Auge nicht quantitativ beurteilen und die heute verwendeten Whole-Slide-Image-Scanner (WSI) erzeugen extrem große Datensätze, was die Bildverarbeitung bei hochauflösenden Bildern mit 85.000 x 40.000 Pixeln oder mehr erschweren kann. Darüber hinaus erfordert die Kommentierung von Bildern, ähnlich wie bei der manuellen Pathologiediagnose, einen erheblichen Zeitaufwand erfahrener Pathologen. Dieser Prozess erschwert die Zusammenstellung einer hochwertigen Datenbank mit beschrifteten Bildern, um ein genaues Diagnosemodell zu trainieren.
Das Computational Digital Pathology Lab (CDPL) am Bioinformatics Institute (BII) von A*STAR hat eine Cloud-basierte Automatisierungsplattform entwickelt, die viele der Herausforderungen im Zusammenhang mit der Deep Learning-gestützten Pathologiediagnose bewältigt und gleichzeitig den Aufwand für Pathologen bei der Bildbeschriftung und klinischen Diagnose reduziert (Abbildung 1). Diese Plattform umfasst A!MagQC, ein vollautomatisches Tool zur Bildqualitätsbewertung, das in MATLAB® mit Deep Learning Toolbox™ und Image Processing Toolbox™ entwickelt wurde. Die Plattform umfasst außerdem ein Deep-Learning-Klassifizierungsmodell, das darauf trainiert ist, Gleason-Muster zu erkennen. In Experimenten mit Pathologen im In- und Ausland reduzierte die Plattform die Bildbeschriftungszeit im Vergleich zur manuellen Annotation und herkömmlichen mikroskopischen Untersuchung um 60 %. So konnten Pathologen Bilder 43 % schneller analysieren und gleichzeitig die gleiche Genauigkeit wie bei herkömmlichen mikroskopischen Untersuchungen beibehalten.
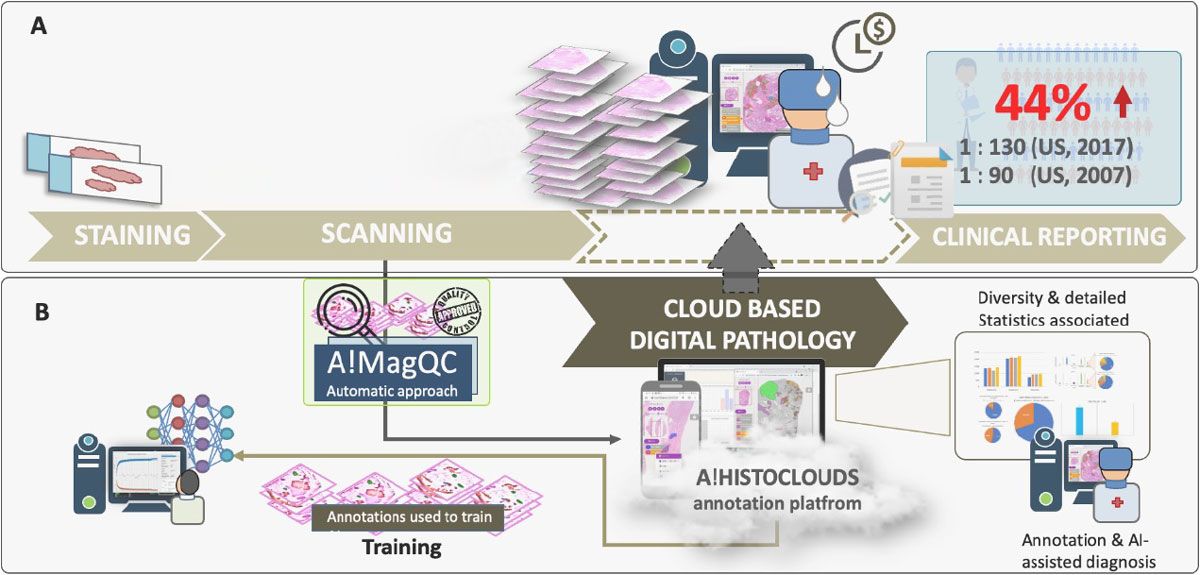
Abbildung 1. Die Plattform zur digitalen Pathologie-Bildanalyse, einschließlich A!MagQC und A!HistoClouds. A) zeigt die vorhandene Pipeline zur digitalen Pathologiebewertung. B) veranschaulicht die in dieser Studie vorgeschlagene Pipeline, die A!MagQC, A!HistoClouds und ein KI-Modell, das Prostatakrebs für von mehreren Scannern gescannte Bilder erkennen und klassifizieren kann, in die vorhandene Pipeline integriert.
Beurteilung der Bildqualität
In der digitalen Pathologie können Bildqualitätsprobleme grob in zwei Kategorien unterteilt werden: Probleme bei der Gewebeprobenvorbereitung und Scanprobleme (Abbildung 2). Geweberisse, Falten, Luftblasen sowie Über- und Unterfärbung fallen in die erste Kategorie. Wenn diese Probleme erkannt werden und die Diagnose beeinträchtigen, muss eine neue Probe vorbereitet werden. Wenn hingegen Probleme mit Bildkontrast, Sättigung oder Fokus festgestellt werden, kann die vorhandene Probe einfach erneut gescannt werden und ein erneutes Schneiden ist nicht erforderlich.

Abbildung 2. Mit A!MagQC erkannte Probleme mit Texturgleichmäßigkeit, Kontrast, Artefakten, Sättigung und Fokus. A) zeigt die einfache und benutzerfreundliche Benutzeroberfläche von A!MagQC. B) zeigt Beispiele für qualitativ minderwertige Bereiche der Ganzfolien-Bilder. C) zeigt die Ausgabe von A!MagQC, wo es in der Lage ist, die Bereiche mit geringer Qualität eines Gesamtbildes in Form von Heatmaps darzustellen.
Unabhängig davon, ob die Analyse von Pathologen oder mithilfe von Deep-Learning-Modellen durchgeführt wird, kann jedes dieser häufigen Probleme nachteilige Auswirkungen haben. Aus diesem Grund hat das BII CDPL-Team von A*STAR Bildverarbeitungsalgorithmen in A!MagQC entwickelt, um die wichtigsten Faktoren, die die Bildqualität beeinflussen, automatisch zu erkennen. Das Team entschied sich für MATLAB, weil es spezialisierte Toolboxen bot. Wenn Bilder beispielsweise zu groß waren, um sie in den Speicher zu laden, könnte die blockproc-Funktion aus der Image Processing Toolbox jedes Bild in Blöcke einer bestimmten Größe aufteilen, diese Block für Block verarbeiten und die Ergebnisse dann zu einem Ausgabebild zusammensetzen.
Das Team verwendete außerdem MATLAB Tools, um die A!MagQC-Benutzeroberfläche zu erstellen und den MATLAB Code zur Verteilung in eine eigenständige ausführbare A!MagQC-Datei zu kompilieren.
Mithilfe der entwickelten QC-Lösung quantifizierte das Team die Bildqualität, um Abweichungen bei Farbe, Helligkeit und Kontrast für WSIs zu ermitteln. Durch diese Übung wurde sichergestellt, dass das anschließend trainierte Deep-Learning-Modell genaue Diagnoseergebnisse für die breite Palette der heute verwendeten Scanner liefert.
Trainieren und Testen von Modellen
Bei der Analyse einer Probe wenden Pathologen das speziell für Prostatakrebs entwickelte Gleason-Bewertungssystem an, um auf Grundlage des Erscheinungsbilds der Probe eine Punktzahl zu vergeben. Neben normalem oder gutartigem Gewebe kann in Teilen der Probe Stroma (Bindegewebe) enthalten sein: Gewebe, dem ein Gleason-Score von 1 bis 5 zugeordnet wird, wobei 5 am bösartigsten ist (Abbildung 3). Bevor das Team mit dem Training eines KI-Diagnosemodells zur Klassifizierung von Gewebeproben beginnen konnte, musste es einen Datensatz aus Bildausschnitten zusammenstellen, die mit diesen Kategorien gekennzeichnet waren. Diese Aufgabe wurde mit Hilfe von Pathologen mithilfe von A!HistoClouds erledigt, die mit Bildern arbeiteten, deren Qualität mit A!MagQC überprüft wurde. Nachdem das Team über einen Basissatz beschrifteter Bildausschnitte verfügte, erweiterte es den Trainingssatz durch eine Datenaugmentation, indem es einzelne Bilder vertikal oder horizontal spiegelte und sie um eine zufällige oder gezielte Gradzahl drehte.

Abbildung 3. Gewebeproben, die Stroma, gutartiges Gewebe und Gewebe mit den Bewertungen Gleason 3, Gleason 4 und Gleason 5 zeigen. Die von Pathologen in A!HistoClouds annotierten Regionen (jeweils mit ihren jeweiligen Kategorien gekennzeichnet) werden als Patches extrahiert. Diese Patches werden für das Modelltraining verwendet.
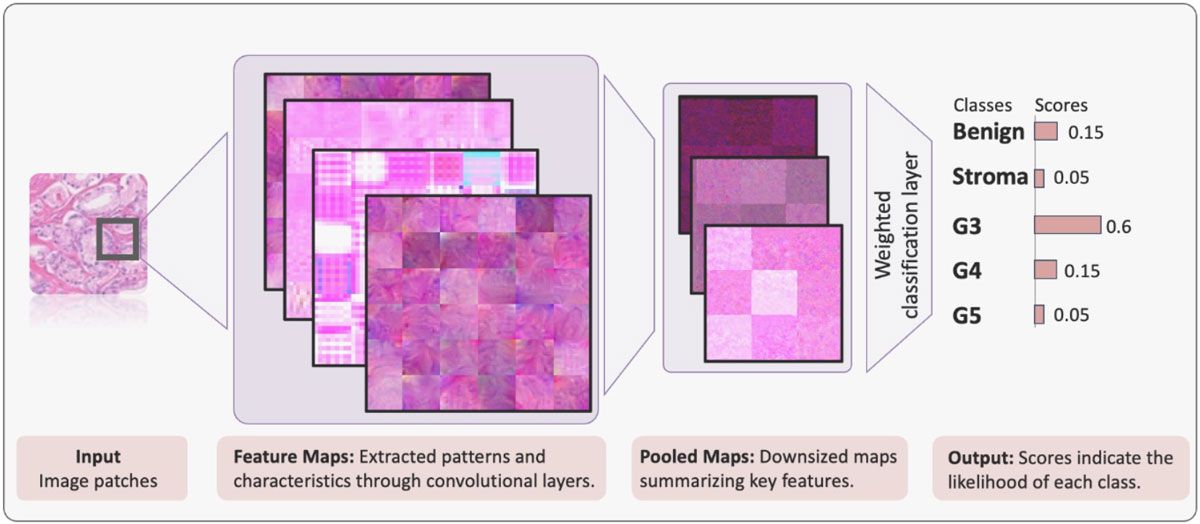
Das Team arbeitete in MATLAB mit Deep Learning Toolbox und erstellte Deep-Learning-Modellstrukturen unter Verwendung der vortrainierten Netzwerke ResNet-50, VGG-16 und NasNet-Mobile und ersetzte deren reguläre Klassifizierungsebenen durch eine gewichtete Klassifizierungsebene (Abbildung 4). Das Team nutzte auch die Option multi-gpu zur Skalierung von einer einzelnen GPU auf mehrere GPUs für das Training des Deep-Learning-Modells.
Das Training und die Anwendung des Modells erfolgen in einem iterativen Prozess. Auf die erste Phase des anfänglichen Trainings anhand manuell beschrifteter Bilder folgt eine zweite, halbautomatische Phase, in der Pathologen die vom trainierten Modell generierten Vorhersagen überprüfen und ändern (Abbildung 5). Diese zweite Phase wird wiederholt, bis das Modell für den Einsatz durch medizinisches Fachpersonal zur Unterstützung klinischer Diagnosen bereit ist. Schritt (a) erfordert eine anfängliche manuelle Annotation durch Pathologen sowohl auf Junior- als auch auf Senior-Ebene. Die Anmerkungen werden mit A!HistoClouds erstellt, wo sie als Patches extrahiert werden, die zum Trainieren des Deep-Learning-Modells verwendet werden. Dieses Modell gibt dann den vorhergesagten Interessenbereich (ROI) aus, um die Pathologen zu unterstützen; daher wird dies als halbautomatische Annotation bezeichnet. In Schritt (b) wird das Modell einem inkrementellen Lernprozess unterzogen. Dabei werden die von der KI vorhergesagten ROIs von Pathologen überprüft und korrigiert, ROIs als Patches extrahiert und das Modell lernt aus diesen neuen Daten. Schritt (b) wird wiederholt, bis die Modellleistung Konvergenz erreicht hat. In Schritt (c) wird das Modell eingesetzt, um eine vollständig automatische Annotation/vollständig automatische Diagnose zu erreichen, die die Entscheidungsfindung der Pathologen unterstützt.
Nächste Schritte
CDPL hat seitdem seine auf Deep Learning basierende Pathologiediagnoseplattform auf globalen Cloudplattformen bereitgestellt und ermöglicht so den in verschiedenen Ländern tätigen Pathologenteams einen einfachen Zugriff. Das BII von A*STAR arbeitet derzeit an der Validierung und Optimierung seines Deep-Learning-Modells für zusätzliche klinische Szenarien, darunter unterschiedliche Gewebedicken, Färbemechanismen und Bildscanner. Schließlich prüft BII Möglichkeiten, denselben Arbeitsablauf zur Bildqualitätsbewertung und zum Deep Learning über Prostatakrebs hinaus auf andere Krebsarten auszuweiten.
CDPL am BII organisierte auch die Automated Gleason Grading Challenge 2022 (AGGC 2022), die auf der International Conference on Medical Image Computing and Computer Assisted Intervention 2022 angenommen wurde. Der Schwerpunkt von AGGC 2022 liegt auf der Bewältigung von Herausforderungen bei der Gleason-Einstufung von Prostatakrebs, der Nutzung digitaler Pathologie und Deep-Learning-Ansätzen. Ziel der Herausforderung ist die Entwicklung automatisierter Algorithmen mit hoher Genauigkeit für H& E-gefärbte histopathologische Bilder der Prostata mit realen Variationen. Dies ist insbesondere die erste Herausforderung im Bereich der digitalen Pathologie, bei der Bildvariationen untersucht und verallgemeinerbare KI-Diagnosemodelle erstellt werden.
Obwohl die Challenge abgeschlossen ist, steht der komplette Datensatz nun für weitere Forschungen zur Verfügung.
Danksagung
Das BII von A*STAR möchte den Kollegen am National University Hospital (NUH), insbesondere Professor Tan Soo Yong und Dr. Susan Hue Swee Shan, Dr. Lau Kah Weng und Dr. Tan Char Loo usw. für ihre Partnerschaft und Zusammenarbeit danken. Die NUH wird gebührend als Quelle der Daten und Proben anerkannt, die zu der in dieser Arbeit beschriebenen Forschung beigetragen haben. Das Team ist dankbar für die Unterstützung durch andere klinische und industrielle Partner.
Veröffentlicht 2024