SimBiology provides apps and programmatic tools for modeling, simulating, and analyzing dynamic systems, focusing on quantitative systems pharmacology (QSP), physiologically-based pharmacokinetic (PBPK), and pharmacokinetic/pharmacodynamic (PK/PD) applications. You can build models interactively using the SimBiology block diagram editor or programmatically using the MATLAB language. Your models can be created from scratch, imported as SBML formatted files, or built on the model examples provided in SimBiology.
SimBiology provides a variety of techniques for analyzing ODE-based models ranging in complexity and size. You can run simulations to assess target feasibility, predict drug efficacy and safety, and identify optimal dosing schedules. You can identify key pathways and parameters using local and global sensitivity analyses and assess biological variability by running parameter sweeps. To estimate parameters you can fit data using nonlinear regression and nonlinear mixed-effects techniques and perform non-compartmental analysis (NCA).
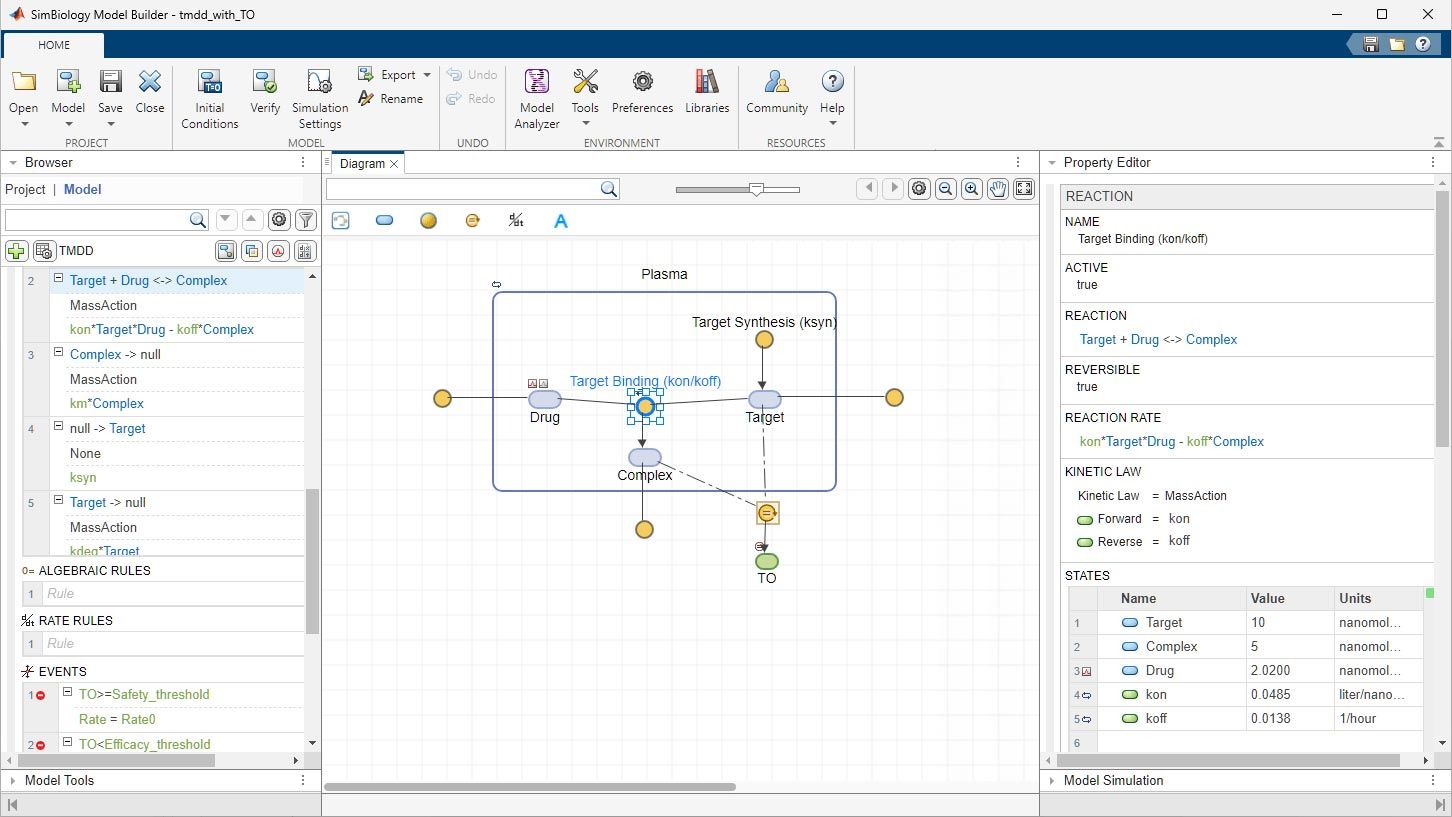
Build QSP, PBPK, and PK/PD Models
Build models just as you would draw them on a piece of paper in SimBiology Model Builder or import existing SBML models. Use dose and variant objects to apply dosing strategies and store alternative quantity values.
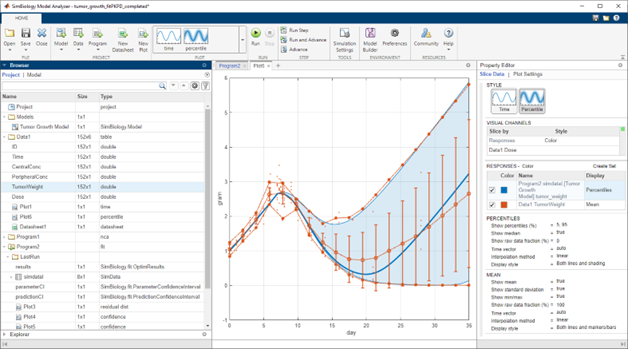
Simulate Model Behavior
Simulate with a variety of deterministic and stochastic solvers using SimBiology Model Analyzer or programmatic tools. Automatic unit conversion brings all quantities into a consistent unit system. Generate reports from analyses.
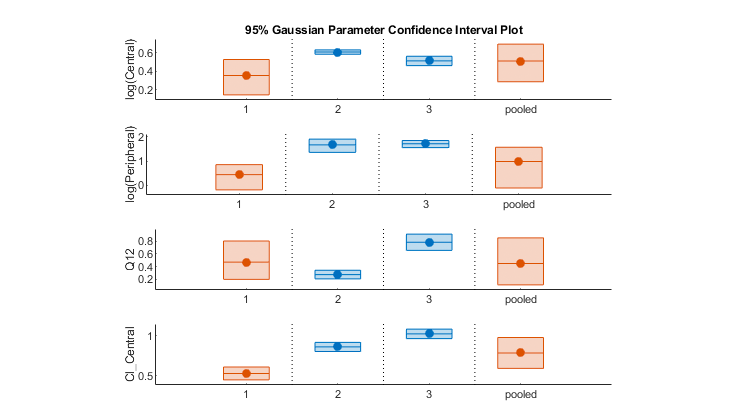
Fit Data to Estimate Parameters
Estimate parameters using nonlinear regression with local, global, or hybrid methods. Calculate parameter and prediction confidence intervals. Account for both fixed and random effects with NLME modeling.
Perform Monte Carlo Simulations
Perform parameter scans and Monte Carlo simulations to assess model behavior under different conditions and dosing regimens. Use interactive sliders to explore how quantity variation impacts model response.
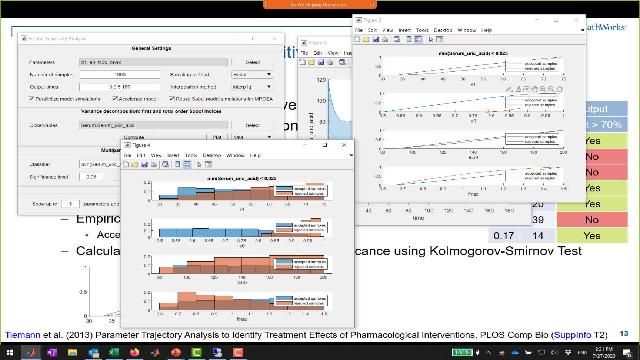
Global and Local Sensitivity Analysis
Explore the effects of variations in model quantities on model response by performing sensitivity analysis. SimBiology supports global, Monte Carlo, and local, derivative-based analyses.
Noncompartmental Analysis
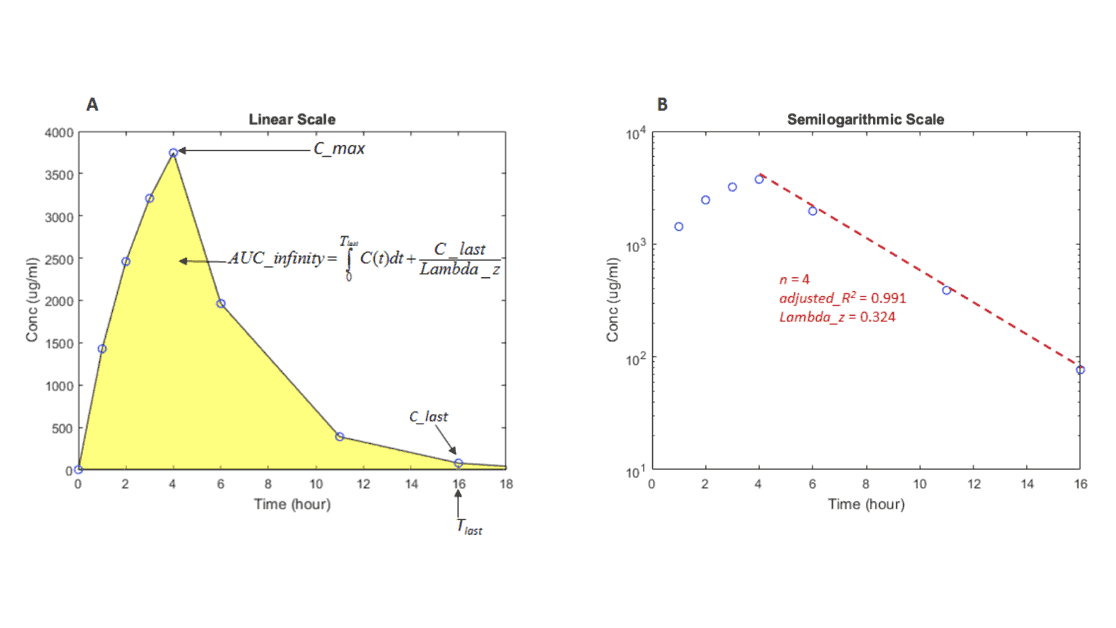
Compute PK parameters from the time course of drug concentrations without assuming a compartmental model. Perform NCA on both experimental and simulation data for single or multiple dosing schemes.
Accelerate Simulations
Accelerate large models or Monte Carlo simulations by converting to compiled C code. Further improve performance by distributing across multiple cores, clusters, or cloud resources with Parallel Computing Toolbox.
Share Models as Web Apps
Create apps in App Designer, package them with MATLAB Compiler, and host them using MATLAB Web App Server. Collaborators can access and run apps in a browser without installing any software.
Community Tools
Use SimBiology programmatically with MATLAB scripts to customize and automate analyses. Community-contributed tools can be used as add-ons for model analysis, such as virtual population simulations.
Product Resources:
Get a Free Trial
30 days of exploration at your fingertips.
Ready to Buy?
Get pricing information and explore related products.
Are You a Student?
Your school may already provide access to MATLAB, Simulink, and add-on products through a campus-wide license.